MYTABOLITES
– un programa de Daryl Lafferty, Arizona State University –
(Idea original: E. Mietzsch, H.T. Lumbsch & J.A. Elix)
[traducción de la pagina: María de los Ángeles (Marusa) Herrera-Campos y Rosa Emilia Pérez-Pérez]
IMPORTANTE !!!
Actualice a la versión más reciente (1.1.2.0). Cuando intenta actualizar la base de datos de la versión 1.0.5.1, el programa devuelve un error y terminará usando un conjunto de datos incompleto [19 de febrero – actualización menor: la versión más nueva ahora es 1.1.2.3, ver más abajo].
Mytaboliteses un programa escrito por Daryl Lafferty en colaboración con Frank Bungartz (Arizona State University), John A. Elix (Australian National University) y Felix Schumm (Wangen) para ayudar en la interpretación de placas de cromatografía de capa fina para el análisis de metabolitos secundarios de líquenes.
Este programa comparte muchas funcionalidades con su antecesor, un software originalmente desarrollado en la Universidad de Essen (Alemania) por Esther Mietzsch- Lumbsch y H. Thorsten Lumbsch, en colaboración con John A. Elix (Australian National University). Los autores nos permitieron generosamente adaptar su idea original para escribir una versión más funcional, la cual está disponible en este portal.
Los datos de la cromatografía incluidos en Mytabolites vienen de la edición más reciente del Catalogue of Standardized Chromatographic Data and Biosynthetic Relationships for Lichen Substances (actualizado regularmente por el autor y en el Consorcio de Herbarios de Líquenes), publicado y distribuido por el autor, J. A. Elix. Los cromatogramas utilizados por el programa fueron publicado originalmente por F. Schumm & J. A. Elix en el Atlas of Images of Thin Layer Chromatograms of Lichen Substances (Books on Demand, 2015) y en el suplemento (Books on Demand, 2016).
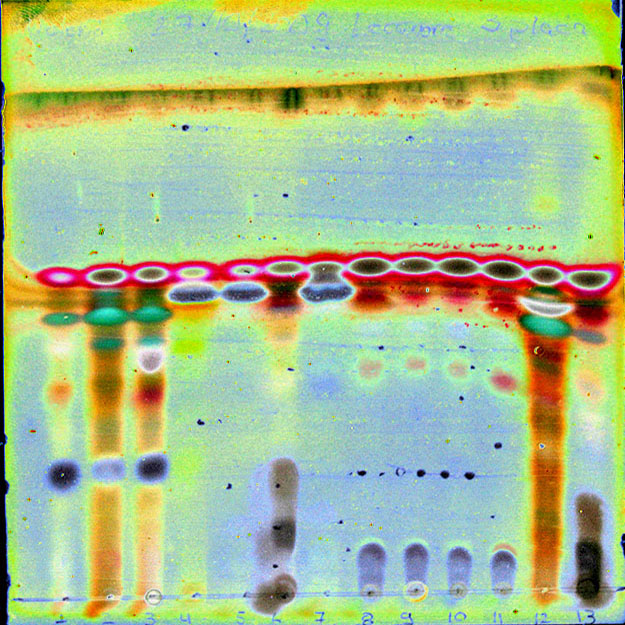
La versión original de Wintabolites fue escrita en Windows de 16 bit. Así que no es compatible con computadoras de 64 bit. Mytabolites solamente corre en Windows (no está disponible para Mac); y actualmente solo existe la versión de Mytabolites en el idioma Inglés. Una alternativa para la identificación de metabolitos secundarios es utilizar NaviKey que no depende del sistema operativo y está disponible como LIASmetabolites en la página del Botanische Staatssammlung München (Alemania).
Todos los datos y cromatogramas también están disponibles en el Glosario del Consorcio de Herbarios de Líquenes.
Actualmente solamente existe la versión de Mytabolites en el idioma inglés.
Para instalar Mytabolites
Para la instalación del programa, hay que crear una carpeta en la unidad de su sistema y descomprimir el archivo Zip. Después se puede ejecutar el programa dando doble-clic en Mytabolites.exe. Si utiliza habitualmente el programa, cree un acceso directo para ejecutar el archivo.
IMPORTANTE:Este archivo es un archivo Zip que incluye un archivo ejecutable (Mytabolites.exe). Por razones de seguridad, muchas instituciones no permiten descargar ni ejecutar este tipo de archivos ejecutables.
Windows 10 y Windows 11 suelen mostrar un mensaje de advertencia de que no se recomienda descargar o ejecutar el programa. Puede ignorar estos mensajes de seguridad, los archivos proporcionados son perfectamente seguros (como lo confirmará cualquier escáner de virus):
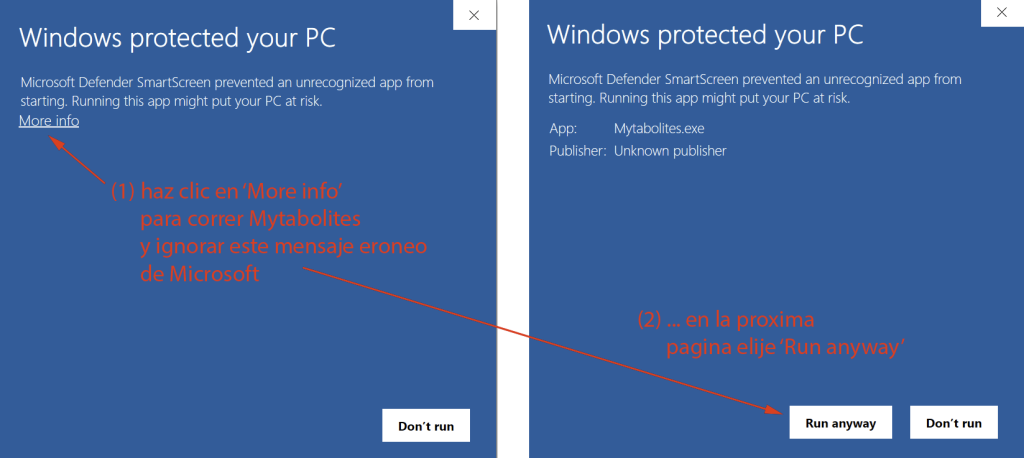
Aun así, es posible que necesite permiso de su institución para instalar/ejecutar el programa. Comuníquese con nosotros en caso de que tenga algún problema (consulte los comentarios a continuación).
Descargar Mytabolites …
Este archivo Zip incluye los siguientes archivos:
- Mytabolites.exe (el archivo de programa ejecutable; ejecute el programa haciendo doble clic o cree un acceso directo del archivo)
- Mytabolites.db [una base de datos, necesario para que el programa acceda a los datos de cromatografía (se puede actualizar desde el Consorcio de Herbarios de Líquenes a través de ‘Sustancias > Actualizar datos de sustancias’)]
- sqlite3.dll (el controlador SQLite es necesario para que el programa puede acceder la base de datos)
- Default.cfg (la configuración de búsqueda predeterminada; no modifique este archivo)
- CurrentSearch.cfg (la configuración de búsqueda personalizada según los parámetros más recientemente especificados
- La primera vez que usted actualice los datos de líquenes y sustancias secundarias desde del Consorcio, también se guardará un archivo de texto llamado UpdateDates.txt en el directorio del programa; cualquier archivo guardado con el programa también se guardará en la misma carpeta.
Cuando ejecute el programa por primera vez, Mytabolites comprobará si hay una versión más nueva disponible y, si está disponible, le solicitará que la instale. También puede buscar actualizaciones manualmente (ver instrucciones de actualización) a través de ‘Help > Help > Program update’.
IMPORTANTE: Con el lanzamiento de Symbiota 3.1, Mytabolites ahora utiliza la interfaz de programación de aplicaciones (API) integrada de Symbiota para actualizar datos del servidor. Mytabolites 1.1.2.0 es más estable y las rutinas de actualización se ejecutan más rápido. También hay algunas correcciones de errores menores y mejoras de funcionalidad. La versión más reciente, 1.1.2.3, es una actualización menor que ahora también incluye una verificación de respaldo, es decir, verifica si la actualización de la base de datos se completó exitosamente y, en caso contrario, restaura la versión anterior de la base de datos..
Mytabolites incluye una base de datos con datos de cromatografía (del catálogo de Elix, es decir, información detallada sobre valores de RF, clases de sustancias, compuestos biosintéticamente relacionados, colores en la placa de TLC antes y después de hornear la placa, reacciones de prueba puntuales, etc.) y datos de líquenes (es decir, qué líquenes contienen metabolitos secundarios particulares según el Consorcio de Herbarios de Líquenes).
Actualizar el Programa:
IMPORTANTE: Si todavía está usando 1.0.5.1. ¡debe actualizar antes de ejecutar cualquier actualización de la base de datos!
En la versión 1.0.5.1. el programa devolverá un error interrumpiendo la actualización. Luego terminará usando un conjunto de datos incompleto (recientemente se lanzó la versión 1.1.2.0 para solucionar este problema. La versión más reciente, 1.1.2.3, es una actualización menor que también incluye una rutina de respaldo, es decir, esta versión verifica si la actualización ha sido exitosa y, si no, restaura la base de datos anterior).
Los datos de cromatografía incluidos en Mytabolites se pueden actualizar desde del Consorcio a través de ‘Substances > Update Substance Data‘ (tenga en cuenta que si cambió manualmente alguna entrada para sustancias particulares usando el botón editar, estos cambios se sobrescribirán cuando actualice los datos con los datos del Consorcio).
La información sobre qué líquenes contienen sustancias particulares, los Datos de Líquenes, se puede actualizar desde del Consorcio a través de ‘Lichens > Update Lichen Data’.
Cada vez que se inicia Mytabolites, el programa comprueba si los datos de cromatografía están actualizados. De lo contrario, se le solicitará hacer la actualización. Si los datos de Liquen tienen más de 80 días, se le pedirá que considere actualizar esa información también:
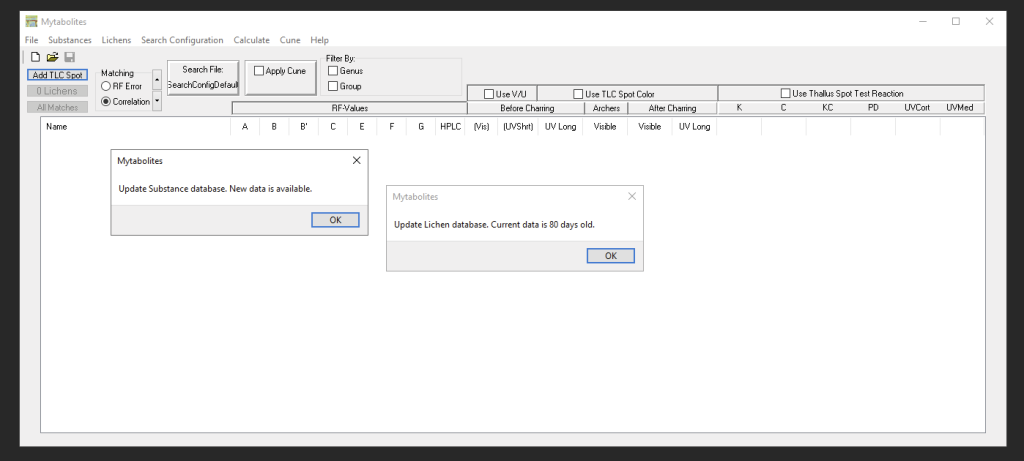
Para obtener información detallada sobre cómo utilizar el programa, consulte el archivo de ayuda integrado en ‘Help > Help’.
Descripción general de la funcionalidad principal:
Ventana principal y algoritmos de coincidencia (configuración de búsqueda)
La ventana principal del programa permite ingresar datos de las manchas de las placas de la TLC procesadas en diferentes solventes a través de ‘Add TLC Spot’. Inmediatamente después de ingresar estos datos, para cada mancha se presenta una lista de posibles coincidencias de metabolitos secundarios. Las manchas se numeran automáticamente como ‘Spot 1’, ‘Spot 2’, etc. Para modificar estos datos haga doble-clic en el nombre de la mancha. Una vez modificados los datos, la lista se actualiza automáticamente. La lista resultante es más y más corta cuanto más información se ingrese (diferentes valores de RF en diferentes solventes, reacciones de color antes y después del horneado de la placa, pruebas de tinción en el talo, etc.):
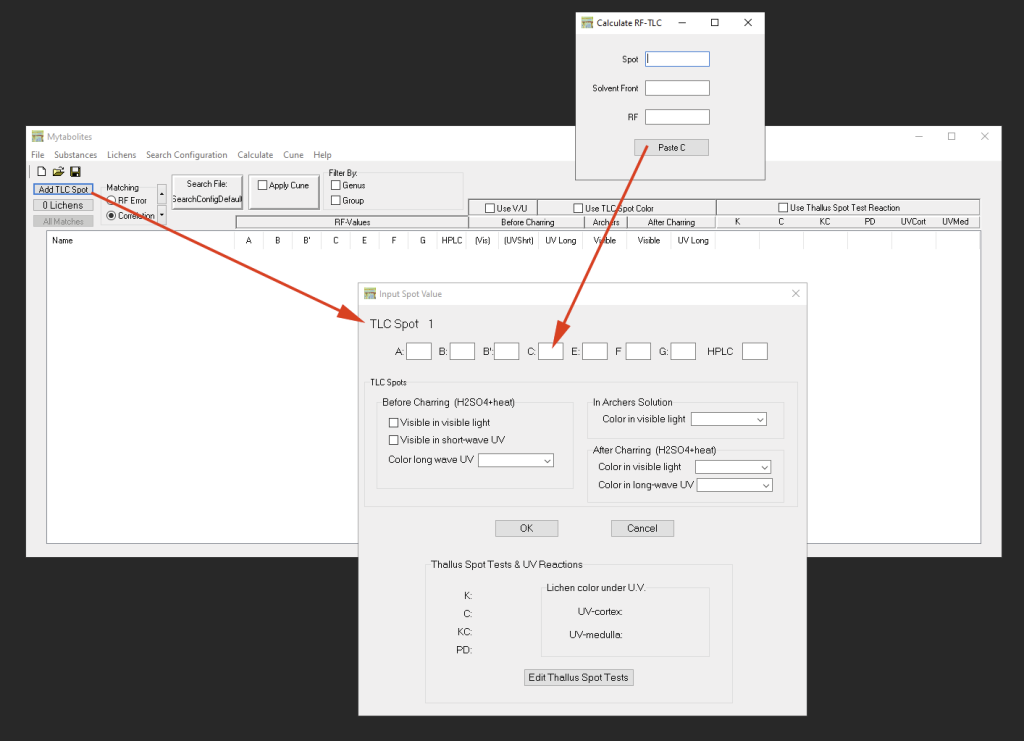
Generalmente, los resultados dependen de los valores de RF de las manchas en la placa de TLC ingresados. La precisión de estas coincidencias se puede mejorar aún más al incluir en los análisis los colores «Before Charring» y «After Charring», tanto en luz visible como en luz ultravioleta. Si las placas fueron tratadas con solución de Archer, ese color también se puede utilizar para obtener resultados más precisos. La lista resultante de las posibles coincidencias se puede reducir aún más ingresando las reacciones de las pruebas de tinción del talo de los líquenes y sus reacciones en luz UV.
Dado que la percepción del color (tanto para las manchas de la placa de TLC como para las pruebas de tinción/reacciones UV del talo) puede ser subjetiva, es posible desactivar los resultados coincidentes con los colores ingresados para ambos.
Cuando se habilita ‘coincidencia de colores’, es posible usar dos algoritmos para el análisis (los criterios se pueden cambiar a través de ‘Search Configuration’):
- RF Error es el algoritmo de coincidencia clásico empleado originalmente por Wintabolites. Este algoritmo simplemente compara el color de las manchas de la placa de TLC con posibles coincidencias incluidas en la base de datos. Dado que la percepción del color es subjetivo, la matriz tolera una cierta cantidad de variación. Por ejemplo, el color «lilac» («lila») también se interpreta como «white» («blanco»), «purple» («morado»), «pink» («rosa»),«bright blue» («azul brillante») y «dark blue»(«azul obscuro»). Al configurar la búsqueda como ‘RF Error’ en el programa simplemente utilice ausencia/presencia de colores con un factor de tolerancia. El valor de este factor es un número entero, normalmente 1 o 2.
- Correlation utilice un algoritmo de coincidencia más «difuso». En ‘Search Configuration’, la cantidad de tolerancia con la que los colores pueden desviarse se puede modificar con doble-clic. El factor de correlación determina cuántas coincidencias aún se toleran. Números más altos permiten más coincidencias. Un factor de correlación de 5 generalmente funciona bien. Aumentar este factor significará que se encontrarán más coincidencias, pero se ordenarán con las mejores coincidencias en la parte superior.
- En ‘SearchConfiguration’ también es posible configurar si las pruebas de tinción y las reacciones UV del talo deben ser consideradas al comparar los datos.
- Con ‘Save As’ es posible guardar las configuraciones de búsqueda individuales. Así es posible re-utilizar diferentes configuraciones de búsqueda para el análisis.
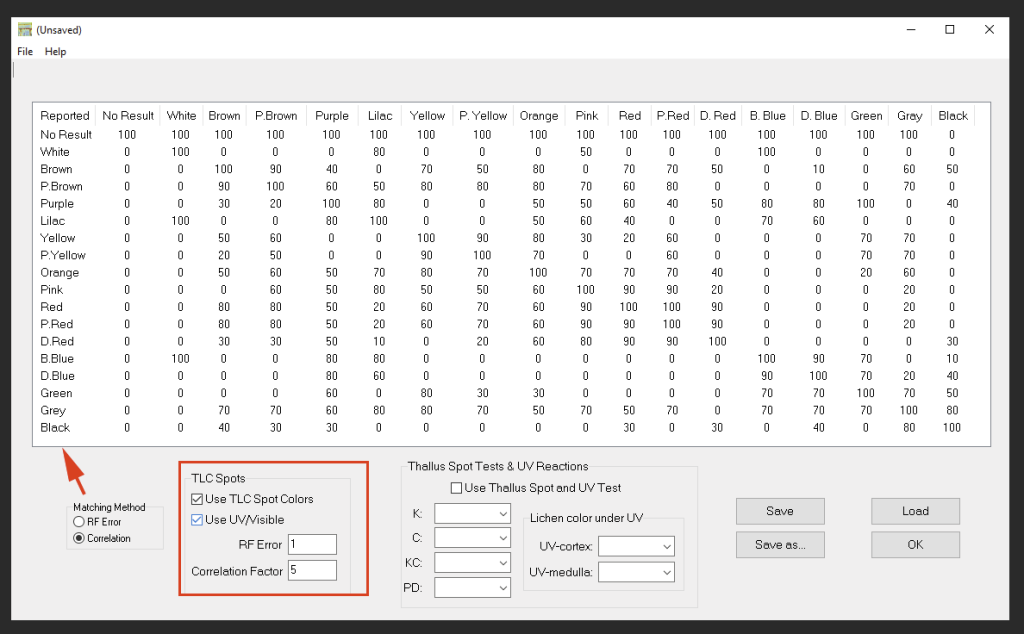
Guardar los resultados
Desde la ventana principal se puede guardar la lista real de las manchas de las placas de TLC como archivos para luego revisar/actualizar estos datos nuevamente. Así, es posible por ejemplo ingresar los valores para un solvente, guardar el archivo y continuar posteriormente, añadiendo información para otros solventes. Al ingresar más información a la lista de resultados, ésta se actualiza automáticamente, pero es necesario guardar estos cambios con ‘Save’.
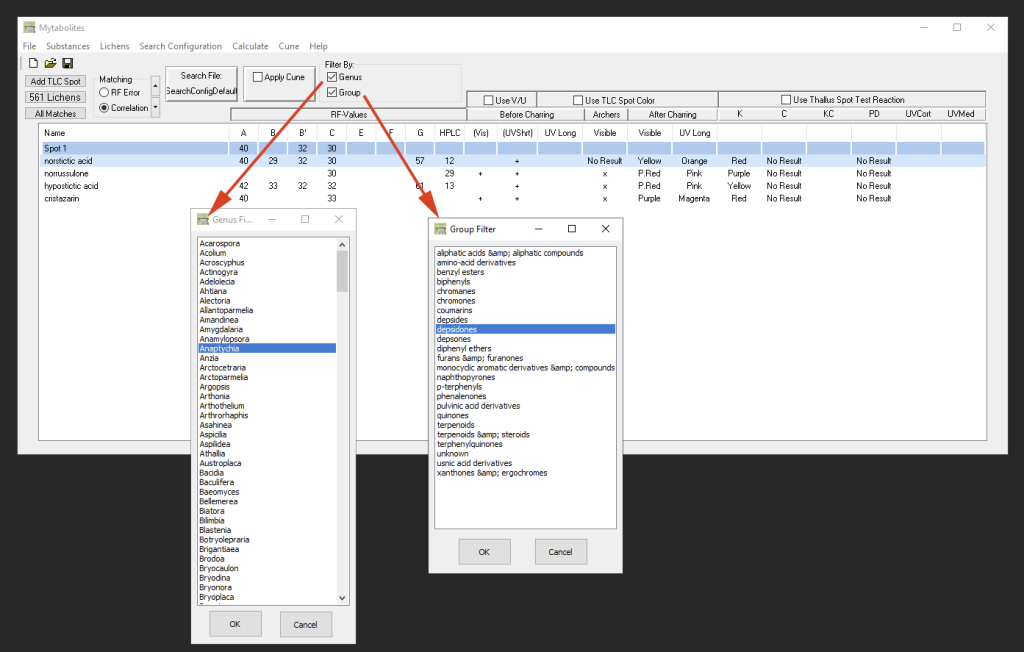
Filtrar resultados de búsqueda
La lista de resultados se puede filtrar por género (metabolitos secundarios reportadas según los datos disponibles en el Consorcio). Otro filtro permite reducir la lista por grupo de sustancias secundarias más probables. Además es posible combinar los dos filtros.
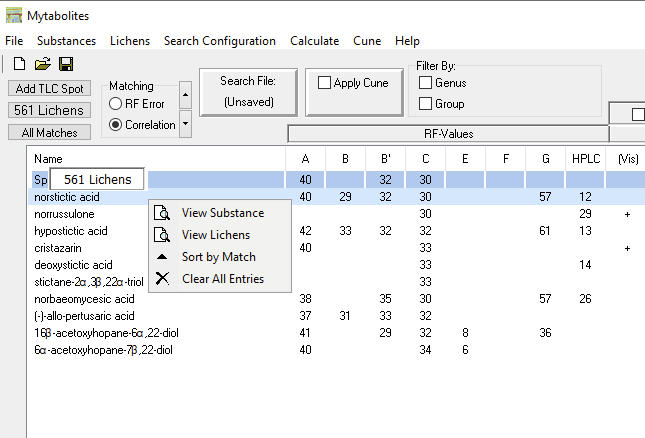
Revisar la lista
Se pueden seleccionar sustancias individuales desde de la lista. Al hacer ‘clic’ con el botón derecho permite explorar los datos cromatográficos detalladas a través de ‘View Substance’. También es posible ver una lista de las especies de líquenes con este metabolito secundario (‘View Lichens’; la cantidad de especies se muestra al pasar el cursor sobre el nombre de la sustancia). Con ‘Sort by Match’ es posible ordenar la lista por coincidencia. Finalmente, el menú permite borrar todos los valores ingresados con ‘Clear All Entries’:
A través de ‘Substances > View/Edit Substances’ se puede acceder y comparar información detallada sobre los metabolitos secundarios (es posible revisar esta base de datos sin ingresar TLC Spots en la ventana principal). Al dar ‘clic’ en el botón derecho, se permite comparar los datos de metabolitos biosintéticamente relacionados:
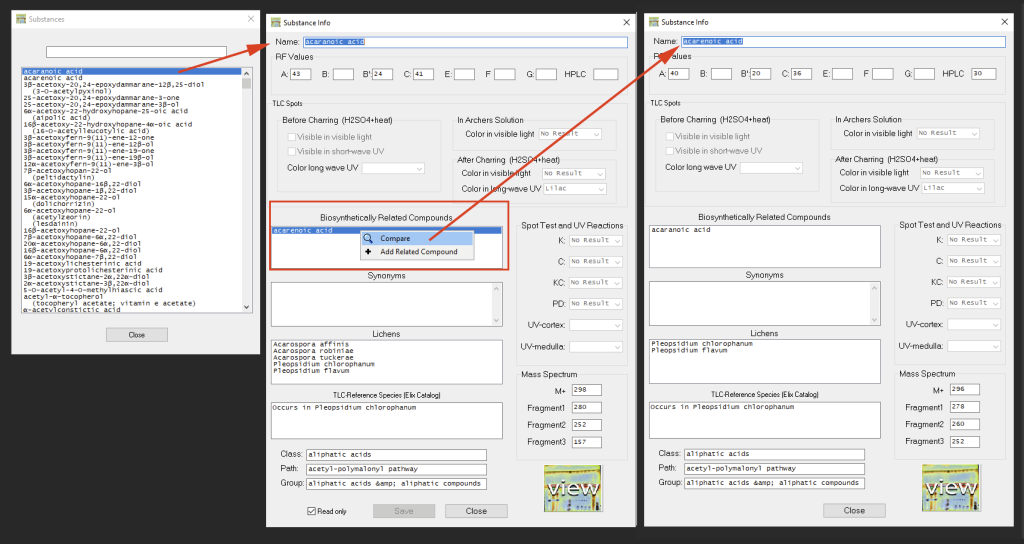
Si existe un cromatograma para una sustancia en particular, se puede usar el botón ‘view’ para mostrarlo (solo funciona cuando el programa tiene acceso a Internet):
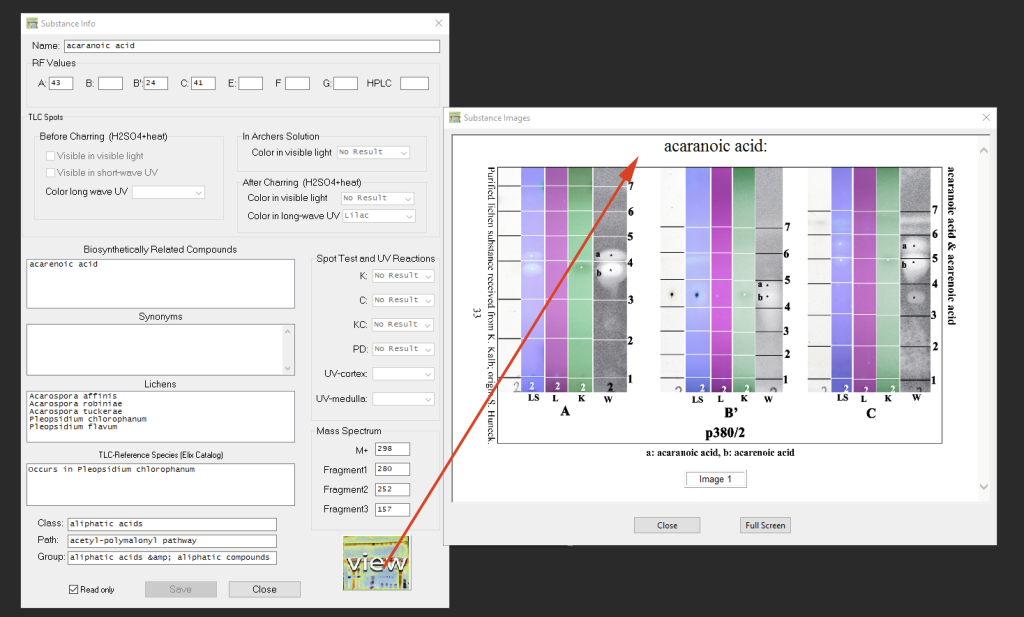
Explorar los datos de metabolitos secundarios:
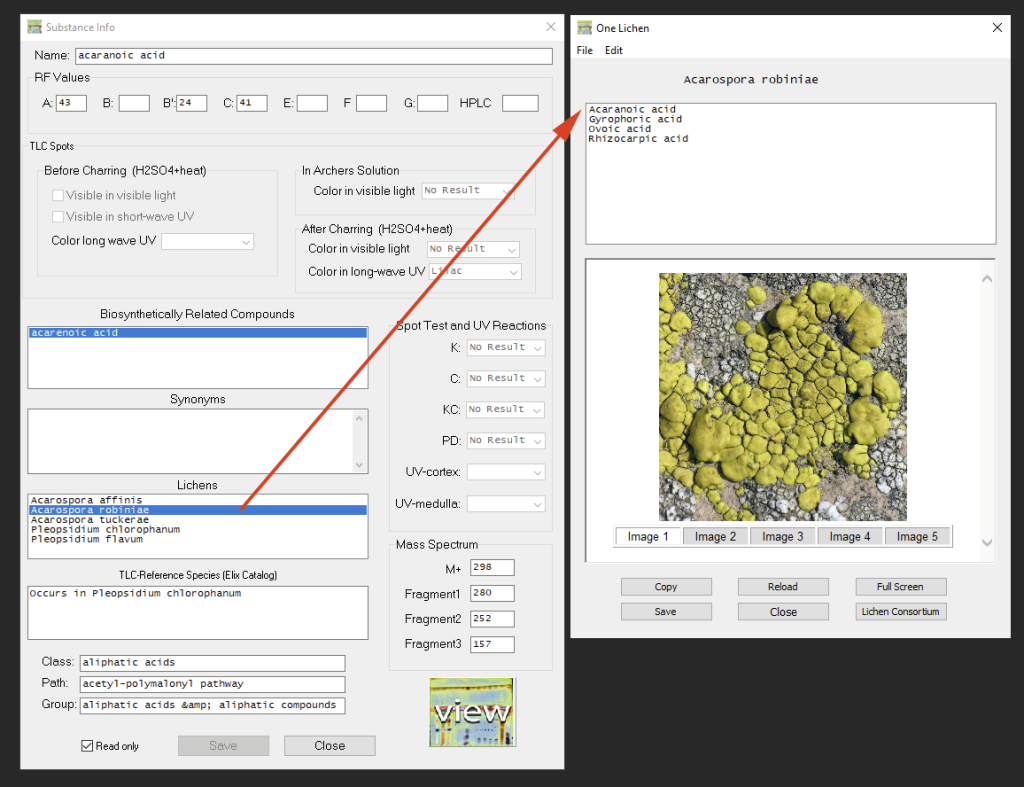
En el Catálogo de Elix se incluye, para cada sustancia, una lista de Especies de Referencia (‘TLC-Reference Species‘) para las cuales se ha reportado dicha sustancia. Además, el programa proporciona una lista de especies de las cuales se ha reportado esta sustancia de acuerdo al Consorcio. Al hacer doble-clic, se accede a la información disponible para dicha especie (las imágenes solo se muestran si existe una conexión a Internet y si hay imágenes disponibles; el botón ‘Lichen Consortium’ abre el perfil del taxón en el navegador de la Internet):
Los datos de las sustancias no se pueden editar a menos que el modo «read-only» esté desactivado (atención: al actualizar los datos del Consorcio, se sobrescribirán todos los cambios manuales; para tener en cuenta la variación en los valores de RF dependiendo de diferentes configuraciones experimentales, se recomienda ajustar los datos a través de ‘Cune’, ver abajo).
Explorar los datos de líquenes:
Es posible revisar la base de datos de líquenes de los cuales se han reportado metabolitos secundarios según el Consorcio. Se puede acceder a esta lista a través de ‘Lichens– View lichens’, con un doble-clic en un nombre en esta lista o utilizando el ‘Search’ para reducir el tamaño de la lista. Desde de la lista de diferentes metabolitos reportados por la especie, es posible revisar los datos de cromatografía dando un doble-clic.
Images of the species are only displayed, if a connection to the internet exists and if an image of that species is available. If available, up to five images can be compared. The button ‘Lichen Consortium’ opens the taxon profile page in the default browser):
Las imágenes de la especie solo se muestran si existe una conexión a Internet y si alguna imagen de esa especie está disponible. Si está disponible en el Consorcio, se pueden comparar hasta cinco imágenes. El botón ‘Lichen Consortium’ abre el perfil del taxón en el navegador de Internet):
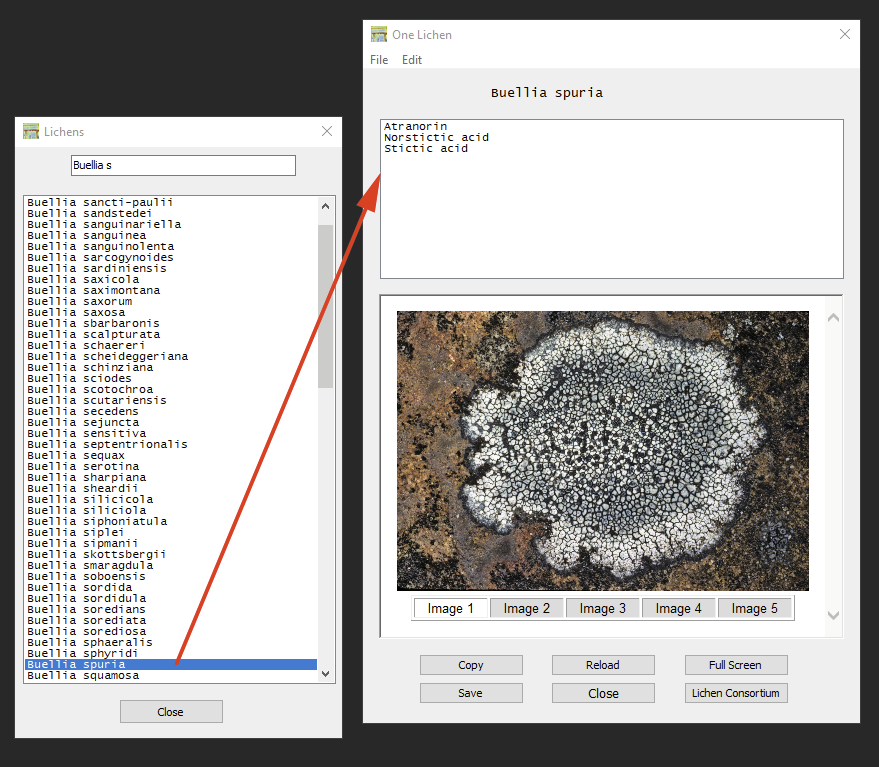
Usando el botón ‘Copy’, se puede copiar la lista de metabolitos secundarios reportados para una especie (los nombres de las sustancias están separados por comas). El botón ‘Save’ creará un archivo de texto de esa lista.
Calcular los valores de RF:
Desde de la ventana principal, el menú ‘Calculate’ abre una calculadora de RF para ingresar la distancia del centro de una mancha de la placa de TLC hasta la línea de inicio al frente del solvente. Esta calculadora también está disponible haciendo doble-clic en el campo de los solventes enumerados en la ventana de ingreso de datos.
Mejorar la precisión y «cunear» (= calibrar) los datos:
Dependiendo de la configuración experimental, es posible que los datos cromatográficos puedan variar. La percepción del color, la lámpara UV utilizada, la cantidad de tiempo y la temperatura a la que han estado expuestas las placas durante el horneado provocan variaciones que afecten la interpretación de los resultados. Modificar la ‘Search Configuration’ para comparar estos resultados con diferentes algoritmos (‘RFError‘, ‘Correlation‘) ayuda con la interpretación de esta variabilidad. Correr placas en diferentes solventes mejora la precisión de los resultados. Es útil consultar la base de datos de líquenes de los que se han reportado metabolitos secundarios específicos, pero estos datos obtenidos desde del Consorcio aún no están completos.
Incluso teniendo en cuenta todos estos aspectos, interpretar los resultados puede seguir siendo difícil, especialmente si la variación experimental causa variación significativa en los valores de RF. Los valores en el catálogo de Elix se basan en una TLC estandarizada, utilizando placas de TLC regulares de 20 x 20 cm, en tanques de vidrio verticales. Los cromatogramas publicados por Schumm & Elix (2015, 2016) corresponden a placas de TLC de 5 x 5 cm. En ASU utilizamos placas de 10 x 10 cm en tanques de TLC de alto rendimiento (HTLC).
Al utilizar configuraciones experimentales tan diferentes, es particularmente importante incluir controles de metabolitos secundarios conocidos para calibrar los valores de RF. Generalmente se recomienda sustancias comunes como la atranorina o el ácido norstictico, pero en teoría es posible utilizar cualquier sustancia conocida. Wintabolites introdujo el concepto de «cuning» de los datos (el término refiere a Bruce McCune de la Oregon State University, quien fue el primero en sugerir este tipo de calibración de valores de RF).
Al utilizar configuraciones experimentales tan diferentes, es muy importante incluir controles de metabolitos secundarios conocidos para calibrar los valores de RF. Generalmente se recomienda usar sustancias comunes como la atranorina o el ácido norestíctico, pero en teoría es posible utilizar cualquier sustancia conocida. Wintabolites introdujo el concepto de «cuning» de los datos (el término hace referencia a Bruce McCune de Oregon State University, quien fue el primero en sugerir este tipo de calibración de valores de RF).
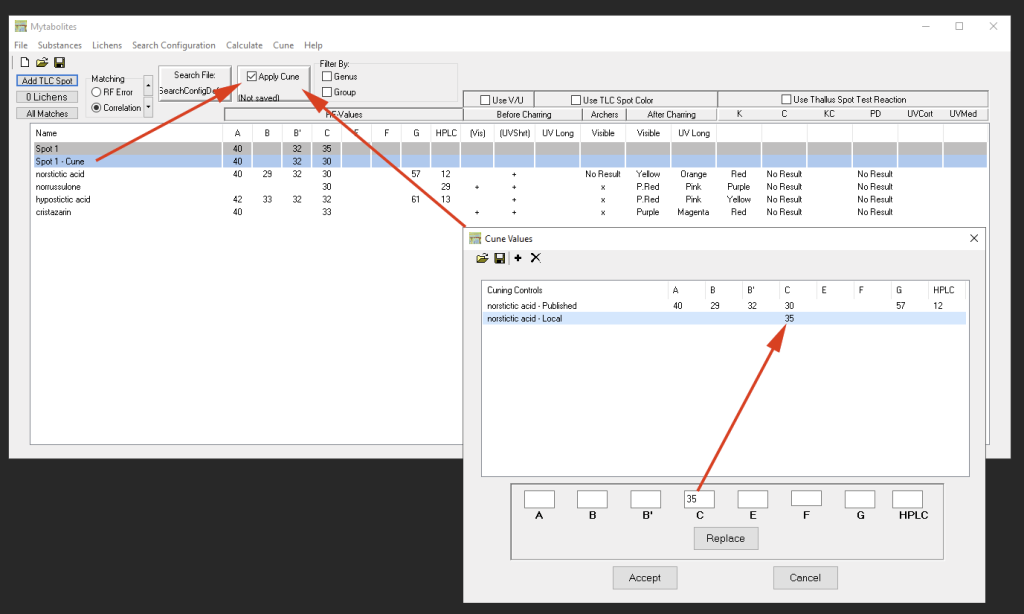
A través de ‘Cune’ se puede añadir una lista de sustancias conocidas (usando el botón ‘+’). Los valores publicados que figuran en el catálogo de Elix se pueden modificar y reemplazarlos con los valores medidos/observados en los diferentes solventes. Con ‘Apply Cune’ en la ventana principal, la lista de resultados se actualizará teniendo en cuenta los valores de RF que se desvían:
Citación:
Para publicaciones, sugerimos citar este programa/sitio web de la siguiente manera:
Lafferty, D., Bungartz, F., Elix, J.A. & Schumm, F. (2024) Mytabolites – a program developed at Arizona State University for the interpretation thin-layer chromatography plates, for the analysis of secondary metabolites from lichens, based on an original concept published by E. Mietzsch, H.T. Lumbsch & J.A. Elix. Help & Resources for the Consortium of Lichen Herbaria, available at https://help.lichenportal.org/index.php/en/resources/metabolites/
Es posible que también desee citar los recursos principales (the Chomatography Catalogue by J.A. Elix and the Chromatogram Atlas by F. Schumm & J.A.Elix) como sigue:
Elix, J.A. (2022) Catalogue of Standardized Chromatographic Data and Biosynthetic Relationships for Lichen Substances. Sixth Edition. Published by the author, Canberra.
Schumm, F. & Elix, J.A. (2015) Atlas of Images of Thin Layer Chromatograms of Lichen Substances. Books on Demand, Norderstedt.
Schumm, F. & Elix, J.A. (2016) Atlas of Images of Thin Layer Chromatograms of Lichen Substances. Supplement. Books on Demand, Norderstedt.
Comentarios:
Agradecemos informes de errores y sugerencias: LichenConsortium@gmail.com
Agradecimientos:
Traducción de la pagina: Dra. María de los Ángeles (‘Marusa’) Herrera-Campos, Dra. Rosa Emilia (‘Rosy’) Pérez-Pérez
